5 Utilisation de ggplot2
5.1 Qu’est ce que ggplot2?
ggplot2 est une librairie développée par Hadley Wickham. Elle permet de réaliser des graphiques élégants et très visuels avec peu de lignes de commande.
La réalisation de graphiques se base sur l’ajout et/ou la modification successives de couches sur un graphique.
Pour l’exemple nous allons utiliser le jeu de données Iris de la librairie datasets. Ce jeu de données regroupe les données morphométriques des sépales et des pétales de 3 espèces d’Iris : Iris setosa, Iris versicolor, Iris virginica.
data(iris) ###chargement du jeu de données iris
summary(iris) ###les paramètres de distribution des variables morphologiques## Sepal.Length Sepal.Width Petal.Length Petal.Width
## Min. :4.300 Min. :2.000 Min. :1.000 Min. :0.100
## 1st Qu.:5.100 1st Qu.:2.800 1st Qu.:1.600 1st Qu.:0.300
## Median :5.800 Median :3.000 Median :4.350 Median :1.300
## Mean :5.843 Mean :3.057 Mean :3.758 Mean :1.199
## 3rd Qu.:6.400 3rd Qu.:3.300 3rd Qu.:5.100 3rd Qu.:1.800
## Max. :7.900 Max. :4.400 Max. :6.900 Max. :2.500
## Species
## setosa :50
## versicolor:50
## virginica :50
##
##
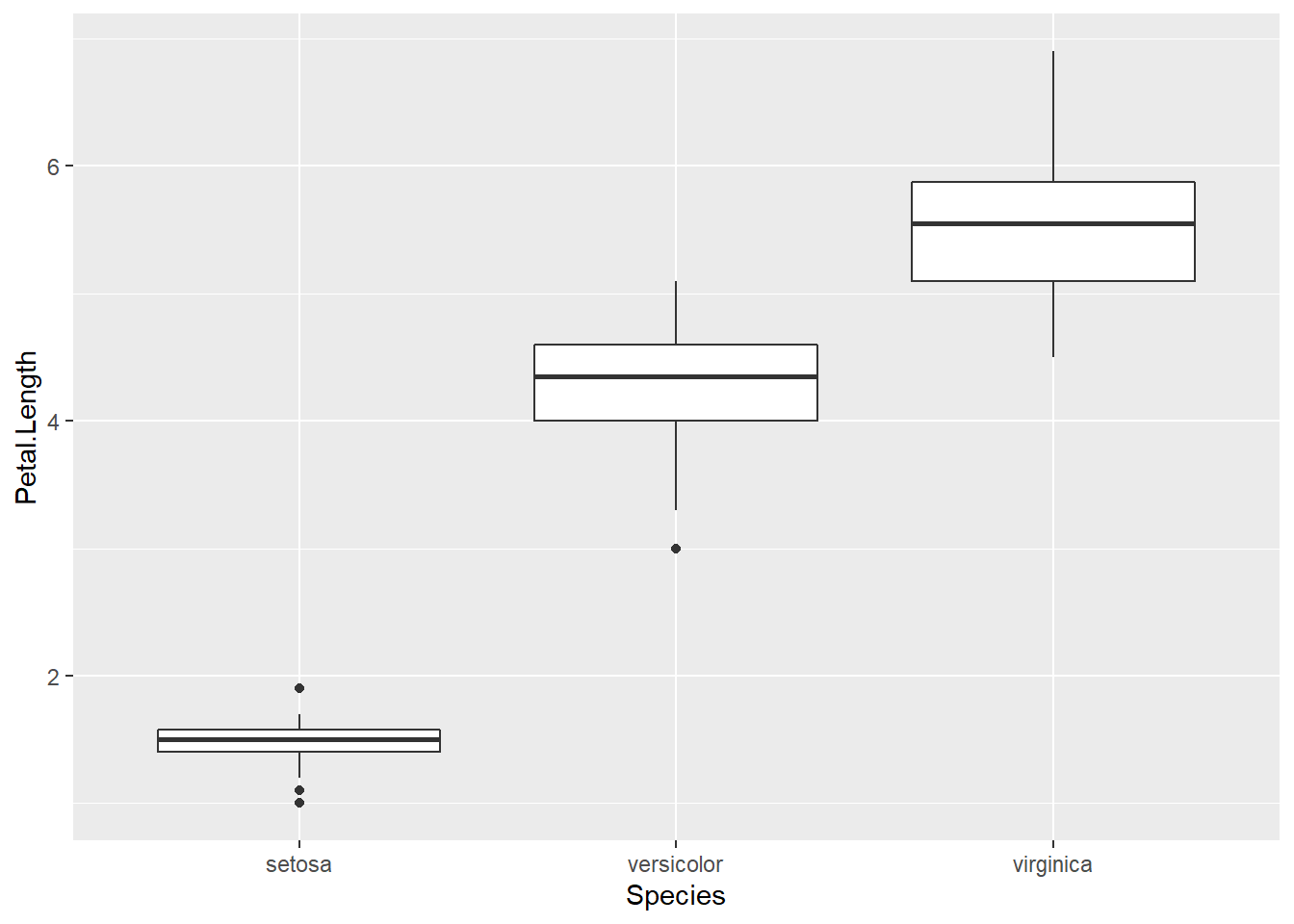
## 5.3 Les boîtes à moustaches
Pour connaitre la distribution d’une variable quantitative entre plusieurs échantillons, vous pouvez utiliser le diagramme à moustache ou boxplot. Dans ggplot2 vous devez utiliser la fonction geom_boxplot().
5.4 Les diagrammes en bâtons
Nous allons calculer la moyenne et l’écart-type de chaque variable quantitative à l’aide la libraire tidyverse également développée par Hadley Wickham.
L’utilisation de la librairie tidyverse sera détaillée dans le TP2.
iris.meansd <-iris %>% group_by(Species) %>% summarise_at(c("Sepal.Length","Sepal.Width","Petal.Length","Petal.Width"),list(mean=mean,sd=sd),na.rm = TRUE)
iris.meansd## # A tibble: 3 x 9
## Species Sepal.Length_me~ Sepal.Width_mean Petal.Length_me~ Petal.Width_mean
## <fct> <dbl> <dbl> <dbl> <dbl>
## 1 setosa 5.01 3.43 1.46 0.246
## 2 versic~ 5.94 2.77 4.26 1.33
## 3 virgin~ 6.59 2.97 5.55 2.03
## # ... with 4 more variables: Sepal.Length_sd <dbl>, Sepal.Width_sd <dbl>,
## # Petal.Length_sd <dbl>, Petal.Width_sd <dbl>## # A tibble: 24 x 3
## Species parameters value
## <fct> <chr> <dbl>
## 1 setosa Sepal.Length_mean 5.01
## 2 versicolor Sepal.Length_mean 5.94
## 3 virginica Sepal.Length_mean 6.59
## 4 setosa Sepal.Width_mean 3.43
## 5 versicolor Sepal.Width_mean 2.77
## 6 virginica Sepal.Width_mean 2.97
## 7 setosa Petal.Length_mean 1.46
## 8 versicolor Petal.Length_mean 4.26
## 9 virginica Petal.Length_mean 5.55
## 10 setosa Petal.Width_mean 0.246
## # ... with 14 more rows## # A tibble: 24 x 4
## Species Variable fonction value
## <fct> <chr> <chr> <dbl>
## 1 setosa Sepal.Length mean 5.01
## 2 versicolor Sepal.Length mean 5.94
## 3 virginica Sepal.Length mean 6.59
## 4 setosa Sepal.Width mean 3.43
## 5 versicolor Sepal.Width mean 2.77
## 6 virginica Sepal.Width mean 2.97
## 7 setosa Petal.Length mean 1.46
## 8 versicolor Petal.Length mean 4.26
## 9 virginica Petal.Length mean 5.55
## 10 setosa Petal.Width mean 0.246
## # ... with 14 more rows## # A tibble: 12 x 4
## Species Variable mean sd
## <fct> <chr> <dbl> <dbl>
## 1 setosa Petal.Length 1.46 0.174
## 2 setosa Petal.Width 0.246 0.105
## 3 setosa Sepal.Length 5.01 0.352
## 4 setosa Sepal.Width 3.43 0.379
## 5 versicolor Petal.Length 4.26 0.470
## 6 versicolor Petal.Width 1.33 0.198
## 7 versicolor Sepal.Length 5.94 0.516
## 8 versicolor Sepal.Width 2.77 0.314
## 9 virginica Petal.Length 5.55 0.552
## 10 virginica Petal.Width 2.03 0.275
## 11 virginica Sepal.Length 6.59 0.636
## 12 virginica Sepal.Width 2.97 0.322Maintenant que nous avons le tableau de données au bon format, nous allons réaliser le diagramme en bâton à l’aide de la fonction geom_bar(). Nous allons également ajouter les barres d’erreurs à l’aide de la fonction geom_errorbar().
graph4 <- ggplot(iris.meansd,aes(x=Species,y=mean,ymin=mean-sd,ymax=mean+sd))+geom_bar(stat="identity")+geom_errorbar()
graph4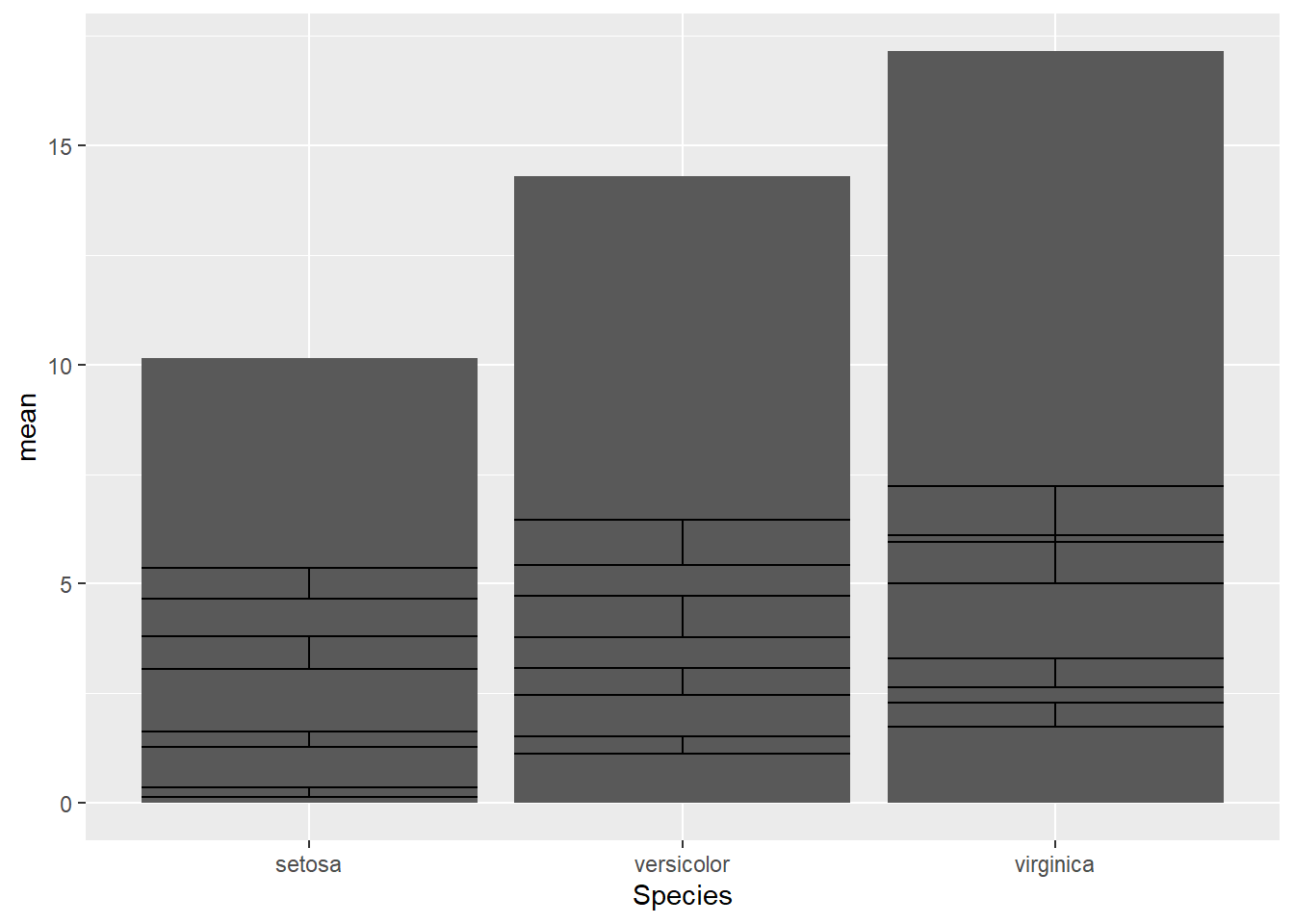
Comme vous pouvez le voir, il y a un problème dans la représentation car nous avons 4 barres d’erreur qui se retrouve dans les bâtons de chaque espèce. Nous avons oublié de réaliser un graphique pour chaque variable "Sepal.Length","Sepal.Width","Petal.Length","Petal.Width". Pour cela, à l’objet graph4nous devons ajouter la fonction facet_grid().
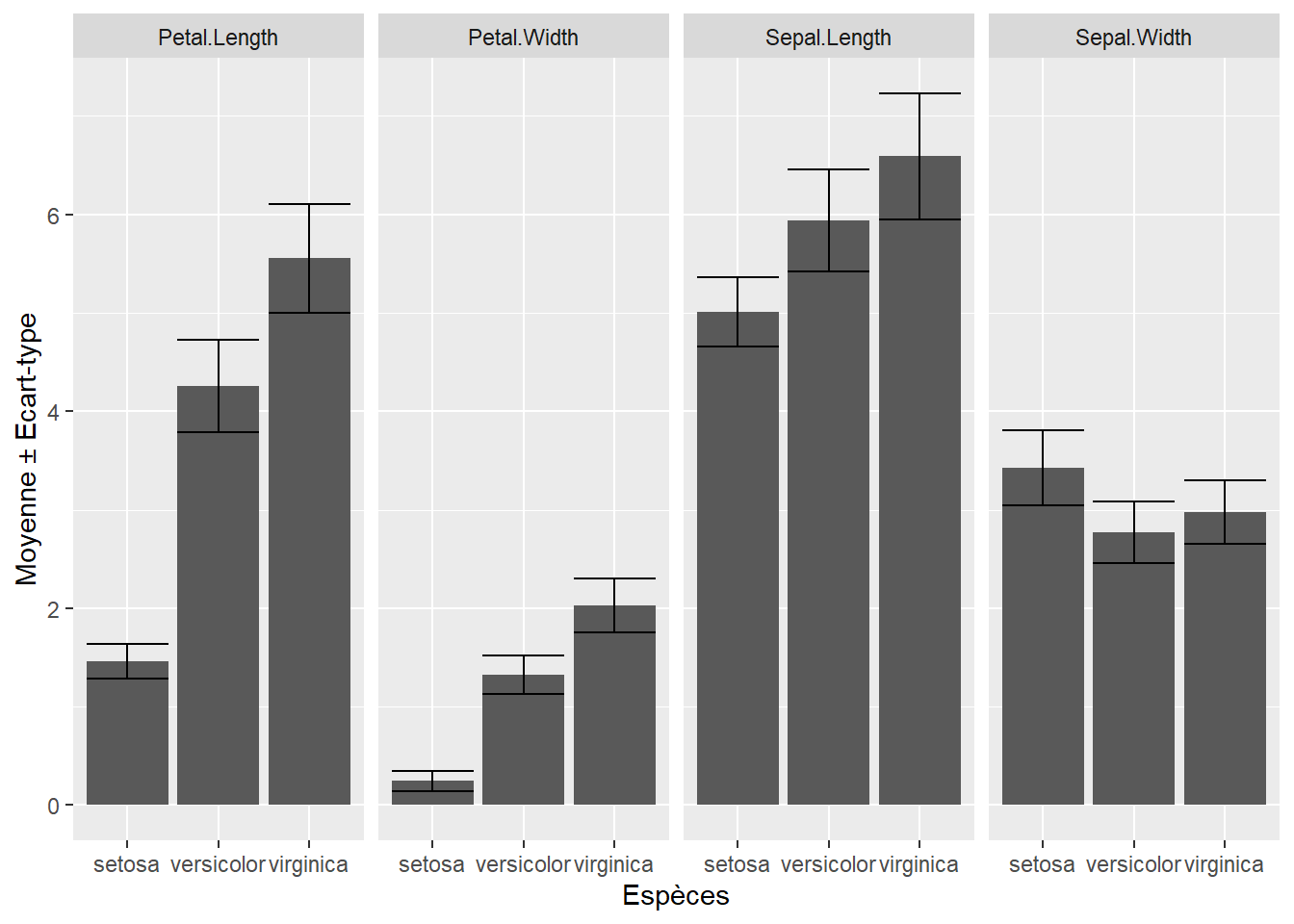
5.2 Comment utiliser
ggplot2?Pour créer un graphique avec
ggplot2, il faut initialiser les paramètres graphiques via la fonctionggplot(). L’orginalité des graphiques deggplot2est qu’ils peuvent être assignés à des objets.5.2.1 Les diagrammes de dispersion
Nous allons réaliser un diagramme de dispersion entre la longueur et la largeur des sépales.
l’argument
aes()correspond aux paramètres esthétiques, on peut définir à l’intérieur les paramètres suivants :x: la variable en abscissey: la variable en ordonnéecolour: couleur des pointspour plus d’information, vous pouvez tapez
?aesOn peut ensuite ajouter la couche graphique correspondant points
geom_point().On va maintenant modifier le titre des axes grâce aux fonctions
xlab()etylab()Le graphique
graph1regroupe les données des trois espèces sans pouvoir les différencier. On va donc différencier les points en fonction des espèces en leur attribuant une couleur. Il faut donc ajouter l’argumentcolourdans les paramètres esthétiques.En utilisant la fonction
facet_grid()on va pouvoir réaliser un graphique pour chaque espèce d’Iris.5.2.2 Les histogrammes
Nous allons réaliser la distribution de fréquences de la longueur des pétales en utilisant la fonction
geom_histogram()Si besoin, nous pouvons changer le nombre de classes pour réaliser un histogramme en modifiant l’argument
bins.