6 Tidyverse - Manipulation de données
Le nom tidyverse de la librairie est une contraction de tidy (“bien rangé”) et de universe. La librairie tidyverse est une collection de librairies ayant une syntaxe cohérente et inter-opérationnelle. Ces librairies ont été en grande partie développées par Hadley Wickham. tidyverse regroupe les librairies suivantes :
ggplot2(visualisation des données)tibble(création de tableaux de données)tidyr(mise en forme)readr(importation des jeux de données)purr(programmation)dplyr(manipulation des données)stringr(chaines de caractères)forcats(variables qualitatives)
Figure 6.1: Librairies présentes dans le tidyverse
Vous pouvez télécharger un aide mémoire en cliquant sur ce lien ou aller sur le (site de tidyverse)
6.1 Les tidy data
Un jeu de données tidy est de la classe tibble suit les principes suivants :
- chaque variable est une colonne
- chaque observation est une ligne
- les lignes n’ont pas de noms
- il n’y a pas de partial matching sur les noms des colonnes
Les tibbles sont compatibles avec les data.frames. La convertion est possible grâce à la fonction as_tibble(). Si le data.frame possède des rownames, il faut les convertir colonnes à l’aide de rownames_to_columns.
Les tableaux tidy sont entièrement compatible avec la librairie ggplot2
6.2 Les pipes
Avec les tidy data, il est possible d’utiliser la fonction pipes %>% qui assigne un tidy à une succession de fonctions de la librairie tidyverse : x %>% f(y) équivaut à f(x,y)
Exemple :
x %>% f(y) %>% g(w) %>% p(l) équivaut à écrire p(g(f(x,y),w),l) ce qui est illisible.
L’utilisation des pipes sera illustrée ci-dessous, mais avant toute chose il faut charger la librairie tidyverse
6.3 dplyr : la librairie pour manipuler les données
6.3.1 slice : sélectionner des lignes
slicesélectionne des lignes du tableau selon leur position
## # A tibble: 1 x 8
## sp sex index FL RW CL CW BD
## <fct> <fct> <int> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 B M 35 16.6 13.5 38.1 43.4 14.9## # A tibble: 10 x 8
## sp sex index FL RW CL CW BD
## <fct> <fct> <int> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 B M 1 8.1 6.7 16.1 19 7
## 2 B M 2 8.8 7.7 18.1 20.8 7.4
## 3 B M 3 9.2 7.8 19 22.4 7.7
## 4 B M 4 9.6 7.9 20.1 23.1 8.2
## 5 B M 5 9.8 8 20.3 23 8.2
## 6 B M 6 10.8 9 23 26.5 9.8
## 7 B M 7 11.1 9.9 23.8 27.1 9.8
## 8 B M 8 11.6 9.1 24.5 28.4 10.4
## 9 B M 9 11.8 9.6 24.2 27.8 9.7
## 10 B M 10 11.8 10.5 25.2 29.3 10.36.3.2 filter : sélectionne par des booléens
Avant de se lancer dans la fonction filter, il faut que l’on voit ensemble les booléens.
Un booléen est une variable de type logique qui a deux états VRAI ou FAUX (TRUE or FALSE). On peut utiliser des booléens pour comparer des valeurs pour cela on utilise les commandes suivantes :
| Conditions initiales | Commandes R | Traduction en Français | Résultats dans R |
|---|---|---|---|
a==b |
a égal à b | FALSE |
|
a!=b |
a différent de b | TRUE |
|
a <- 1 |
a>b |
a strictement supérieur à b | FALSE |
b <- 3 |
a>=b |
a supérieur ou égal b | FALSE |
a<b |
a strictement inférieur à b | TRUE |
|
a<=b |
a inférieur ou égale à b | TRUE |
|
a <- 1 |
a==b & c>b |
a égal à b ET c strictement supérieur à b | FALSE |
b <- 3 |
a==b | c>b |
a égal à b OU c strictement supérieur à b | TRUE |
c <- 4 |
a<=b & c>b |
a inférieur ou égale à b ET c strictement supérieur à b | TRUE |
a>=b | c<b |
a supérieur ou égale à b OU c strictement inférieur à b | FALSE |
Revenons maintenant à la fonction filter qui sélectionne des lignes selon une condition et n’affiche que les lignes dont le résultat de la condition est TRUE. Dans l’exemple ci-dessous, nous allons sélectionner uniquement les crabes qui sont oranges (sp=="O")
## # A tibble: 100 x 8
## sp sex index FL RW CL CW BD
## <fct> <fct> <int> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 O M 1 9.1 6.9 16.7 18.6 7.4
## 2 O M 2 10.2 8.2 20.2 22.2 9
## 3 O M 3 10.7 8.6 20.7 22.7 9.2
## 4 O M 4 11.4 9 22.7 24.8 10.1
## 5 O M 5 12.5 9.4 23.2 26 10.8
## 6 O M 6 12.5 9.4 24.2 27 11.2
## 7 O M 7 12.7 10.4 26 28.8 12.1
## 8 O M 8 13.2 11 27.1 30.4 12.2
## 9 O M 9 13.4 10.1 26.6 29.6 12
## 10 O M 10 13.7 11 27.5 30.5 12.2
## # ... with 90 more rowsLes crabes femelles :
## # A tibble: 100 x 8
## sp sex index FL RW CL CW BD
## <fct> <fct> <int> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 B F 1 7.2 6.5 14.7 17.1 6.1
## 2 B F 2 9 8.5 19.3 22.7 7.7
## 3 B F 3 9.1 8.1 18.5 21.6 7.7
## 4 B F 4 9.1 8.2 19.2 22.2 7.7
## 5 B F 5 9.5 8.2 19.6 22.4 7.8
## 6 B F 6 9.8 8.9 20.4 23.9 8.8
## 7 B F 7 10.1 9.3 20.9 24.4 8.4
## 8 B F 8 10.3 9.5 21.3 24.7 8.9
## 9 B F 9 10.4 9.7 21.7 25.4 8.3
## 10 B F 10 10.8 9.5 22.5 26.3 9.1
## # ... with 90 more rowsLes crabes mâles ET bleus :
## # A tibble: 50 x 8
## sp sex index FL RW CL CW BD
## <fct> <fct> <int> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 B M 1 8.1 6.7 16.1 19 7
## 2 B M 2 8.8 7.7 18.1 20.8 7.4
## 3 B M 3 9.2 7.8 19 22.4 7.7
## 4 B M 4 9.6 7.9 20.1 23.1 8.2
## 5 B M 5 9.8 8 20.3 23 8.2
## 6 B M 6 10.8 9 23 26.5 9.8
## 7 B M 7 11.1 9.9 23.8 27.1 9.8
## 8 B M 8 11.6 9.1 24.5 28.4 10.4
## 9 B M 9 11.8 9.6 24.2 27.8 9.7
## 10 B M 10 11.8 10.5 25.2 29.3 10.3
## # ... with 40 more rowsSi vous souhaitez filter plusieurs variables en même temps vous pouvez utiliser la fonction de la manière suivante : filter(cond1, cond2, cond3, ..., condn). Attention, cette manière d’écrire équivaut à utiliser la fonction logique ET (&), ce qui signifie que la totalité des conditions doivent être vraies pour que la sélection soit vraie.
6.3.3 select : sélectionne des colonnes
selectpermet de selectionner des colonnes.
## # A tibble: 200 x 3
## sex CL CW
## <fct> <dbl> <dbl>
## 1 M 16.1 19
## 2 M 18.1 20.8
## 3 M 19 22.4
## 4 M 20.1 23.1
## 5 M 20.3 23
## 6 M 23 26.5
## 7 M 23.8 27.1
## 8 M 24.5 28.4
## 9 M 24.2 27.8
## 10 M 25.2 29.3
## # ... with 190 more rowsselect intègre des fonctions falicitant la sélection de colonnes.
starts_with()ends_with()contains
6.3.4 mutate création de nouvelles colonnes
mutate permet de créer de nouvelles colonnes à partir de colonnes existantes.
## # A tibble: 200 x 2
## sex form
## <fct> <dbl>
## 1 M 0.847
## 2 M 0.870
## 3 M 0.848
## 4 M 0.870
## 5 M 0.883
## 6 M 0.868
## 7 M 0.878
## 8 M 0.863
## 9 M 0.871
## 10 M 0.860
## # ... with 190 more rows6.3.5 group_by : regroupement
group_by permet de définir des groupes de lignes à partir des vlaeurs d’une ou plusieurs colonnes.
## # A tibble: 200 x 8
## # Groups: sex [2]
## sp sex index FL RW CL CW BD
## <fct> <fct> <int> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 B M 1 8.1 6.7 16.1 19 7
## 2 B M 2 8.8 7.7 18.1 20.8 7.4
## 3 B M 3 9.2 7.8 19 22.4 7.7
## 4 B M 4 9.6 7.9 20.1 23.1 8.2
## 5 B M 5 9.8 8 20.3 23 8.2
## 6 B M 6 10.8 9 23 26.5 9.8
## 7 B M 7 11.1 9.9 23.8 27.1 9.8
## 8 B M 8 11.6 9.1 24.5 28.4 10.4
## 9 B M 9 11.8 9.6 24.2 27.8 9.7
## 10 B M 10 11.8 10.5 25.2 29.3 10.3
## # ... with 190 more rowsSi on applique la fonction slice sur un tableau groupé, la fonction va sélectionner les lignes aux positions indiqués pour chaque groupe.
## # A tibble: 2 x 8
## # Groups: sex [2]
## sp sex index FL RW CL CW BD
## <fct> <fct> <int> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 B F 1 7.2 6.5 14.7 17.1 6.1
## 2 B M 1 8.1 6.7 16.1 19 76.3.6 summarise : réaliser un résumé
summarisepermet de résumer un tableau en effectuant 1 ou plusieurs opérations.
## # A tibble: 2 x 3
## sex CL.mean CW.mean
## <fct> <dbl> <dbl>
## 1 F 31.4 35.8
## 2 M 32.9 37.06.3.7 tally : tableau de contingence
tally permet de compter le nombre d’observations d’un groupe
## # A tibble: 4 x 3
## # Groups: sex [2]
## sex sp n
## <fct> <fct> <int>
## 1 F B 50
## 2 F O 50
## 3 M B 50
## 4 M O 50###arrange : réordonne un tableau
La fonction arrange() ordonne les lignes en fonction des valeurs des variables sélectionnées. Dans l’exemple ci-dessous, nous allons ordonner les lignes du tableau en rangeant par ordre croissant les valeurs de la variable CL.
## # A tibble: 200 x 8
## sp sex index FL RW CL CW BD
## <fct> <fct> <int> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 B F 1 7.2 6.5 14.7 17.1 6.1
## 2 B M 1 8.1 6.7 16.1 19 7
## 3 O M 1 9.1 6.9 16.7 18.6 7.4
## 4 B M 2 8.8 7.7 18.1 20.8 7.4
## 5 B F 3 9.1 8.1 18.5 21.6 7.7
## 6 B M 3 9.2 7.8 19 22.4 7.7
## 7 B F 4 9.1 8.2 19.2 22.2 7.7
## 8 B F 2 9 8.5 19.3 22.7 7.7
## 9 B F 5 9.5 8.2 19.6 22.4 7.8
## 10 B M 4 9.6 7.9 20.1 23.1 8.2
## # ... with 190 more rowsSi vous souhaitez ranger les valeurs de la variable CL par ordre décroissant, il faudrat utiliser la fonction desc() (descending) :
## # A tibble: 200 x 8
## sp sex index FL RW CL CW BD
## <fct> <fct> <int> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 O M 50 23.1 15.7 47.6 52.8 21.6
## 2 O M 49 23 16.8 47.2 52.1 21.5
## 3 B M 50 21.3 15.7 47.1 54.6 20
## 4 O F 50 23.1 20.2 46.2 52.5 21.1
## 5 O M 45 21.6 15.4 45.7 49.7 20.6
## 6 O M 44 21.5 15.5 45.5 49.7 20.9
## 7 O M 47 21.9 15.7 45.4 51 21.1
## 8 O M 48 22.1 15.8 44.6 49.6 20.5
## 9 O F 45 21.3 18.4 43.8 48.4 20
## 10 O M 46 21.6 14.8 43.4 48.2 20.1
## # ... with 190 more rowsIl est également possible de ranger les lignes du tableau en utilisant plusieurs variables :
## # A tibble: 200 x 8
## sp sex index FL RW CL CW BD
## <fct> <fct> <int> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 O M 50 23.1 15.7 47.6 52.8 21.6
## 2 O M 49 23 16.8 47.2 52.1 21.5
## 3 B M 50 21.3 15.7 47.1 54.6 20
## 4 O F 50 23.1 20.2 46.2 52.5 21.1
## 5 O M 45 21.6 15.4 45.7 49.7 20.6
## 6 O M 44 21.5 15.5 45.5 49.7 20.9
## 7 O M 47 21.9 15.7 45.4 51 21.1
## 8 O M 48 22.1 15.8 44.6 49.6 20.5
## 9 O F 45 21.3 18.4 43.8 48.4 20
## 10 O M 46 21.6 14.8 43.4 48.2 20.1
## # ... with 190 more rowsLorsque l’on souhaite arranger des lignes d’un tableau en fonction de groupes (group_by()), il faut spécifier l’argument .by_group = TRUE.
## # A tibble: 200 x 8
## # Groups: sex [2]
## sp sex index FL RW CL CW BD
## <fct> <fct> <int> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 O F 50 23.1 20.2 46.2 52.5 21.1
## 2 O F 45 21.3 18.4 43.8 48.4 20
## 3 O F 49 22.5 17.2 43 48.7 19.8
## 4 O F 48 21.9 17.2 42.6 47.4 19.5
## 5 O F 47 21.7 17.1 41.7 47.2 19.6
## 6 O F 43 20.6 17.5 41.5 46.2 19.2
## 7 O F 46 21.4 18 41.2 46.2 18.7
## 8 B F 50 19.2 16.5 40.9 47.9 18.1
## 9 O F 39 20 16.7 40.4 45.1 17.7
## 10 O F 38 19.9 17.9 40.1 46.4 17.9
## # ... with 190 more rows6.3.8 bind_rows et bind_cols : assemblage de tableaux
La fonction bind_rows permet d’assembler des tableaux par les lignes et bind_cols par les colonnes.
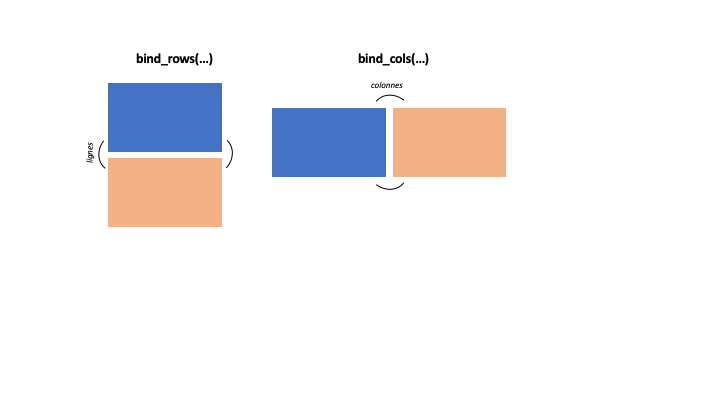
Figure 6.2: Schéma explicatif des fonctions bind_rows et bind_cols
6.3.9 inner_join-full_join-lef_join-… : joindre des tableaux
Contrairement aux deux fonctions précédentes (section 6.3.8), les fonctions _join nécessitent que les tableaux aient une ou plusieurs colonnes communes pour réaliser une jointure. L’utilisation des différentes fonctions de jointures sont décrites dans la figure 6.3
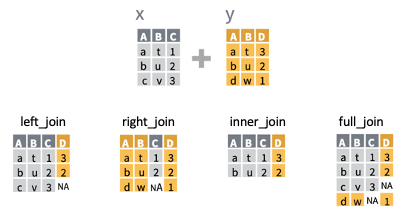
Figure 6.3: Schéma explicatif des fonctions bind_rows et bind_cols
left_join: jointure des valeurs du tableau Y au tableau Xright_join: jointure des valeurs du tableau X au tableau Yinner_join: Seules les lignes et les colonnes présentent dans X et Y sont jointesfull_join: Toutes les valeurs des tableaux X et Y sont jointes
Si vous souhaitez compléter vos connaissances vous pouvez consulter un aide mémoire en cliquant sur ce lien ou aller sur le (site de tidyverse)